前言
定义
单细胞测序方法即single cell sequencing(SNS),能准确定量一个单细胞核中基因拷贝数目。由于癌细胞中基因组部分被删除,或者扩增,从而引起关键基因的缺失,或者表达过量,干扰正常细胞生长,因此利用这种方法就能分析基因拷贝数目,从而诊断癌症。
关于Scanpy
Scanpy是一种可扩展的工具包,用于分析与Anndata共同构建的单细胞基因表达数据。它包括预处理,可视化,聚类,轨迹推理和差异表达测试。基于Python的实施有效地处理了超过一百万个单元的数据集。
环境配置
安装anaconda3
首先安装Anaconda3,Anaconda自带的软件包太多太杂,有一些工具并不需要,对于想最快速部署的玩家可以安装miniconda或mini-forge。不过建议初学者还是使用Anaconda较为方便。
下载地址:
设置conda-forge为优先频道
- 安装好了之后,打开Anaconda Promot(Powershell或者cmd都可以)

- 输入
conda config --set show_channel_urls yes - 到用户根部目录下找到.condarc文件,打开
- 将配置文件修改为以下
channel_priority: strict
channels:
- conda-forge
- defaults
show_channel_urls: true
ssl_verify: false
这样默认安装的包就会都从conda-forge通道下载了(这很重要,可以避免不必要的依赖冲突)
建立虚拟环境
- 还是打开Anaconda Promot(Powershell或者cmd都可以)

- 输入
conda create --name scanpy python=3.10创建名为scanpy,python版本为3.10的虚拟环境 - 输入
conda activate scanpy激活刚刚创建的新环境,这时候安装的包才会装入这个新建立的环境中 - 为什么要这样?据说为了具体用途只配置相应的包而不在一个环境中来个大杂烩可以显著提高性能以及加载速度。
安装scanpy
参考官方文档Scanpy
-
激活环境
conda activate scanpy -
输入
conda install scanpy python-igraph leidenalg
此处官方文档是conda install -c conda-forge scanpy python-igraph leidenalg, 由于我们前已经设置conda-forge为优先频道所以就不必加-c强制使用conda-forge频道了。
到这里,scanpy环境就已经配置好了。不过,如果要更舒适地编辑环境,还需要安装ipykernel来让jupyter notebook(Anaconda中附带安装的Web编辑器,优点挺多可以自行百度搜索)可以识别到这个新建立的环境
- 输入
conda install ipykernel - 然后输入
python -m ipykernel install --name scanpy - 如果想在VSCode里使用jupyter还必须再装一遍ipywidgets、ipython才能正常工作
conda install ipython ipywidgets
开始
好了,让我们进入jupyter notebook(又或者VSCode)吧
- Jupyter notebook
(base) PS C:\Users\yzgop> conda activate scanpy
(scanpy) PS C:\Users\yzgop> jupyter notebook
点击New新建ipynb选择scanpy这个环境
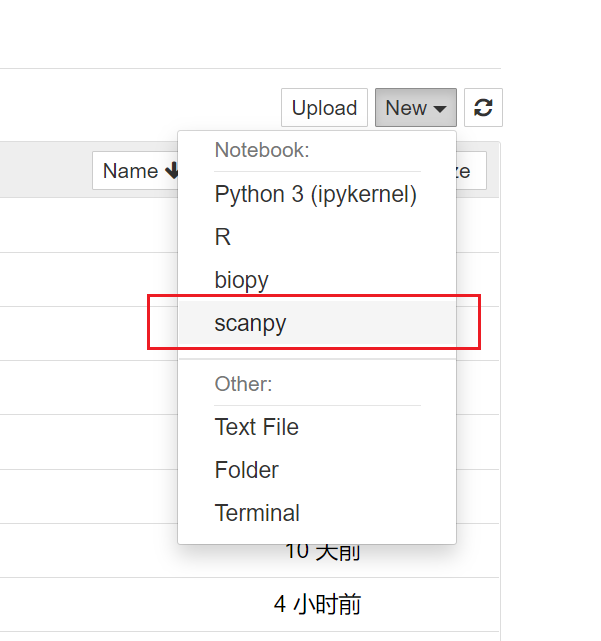
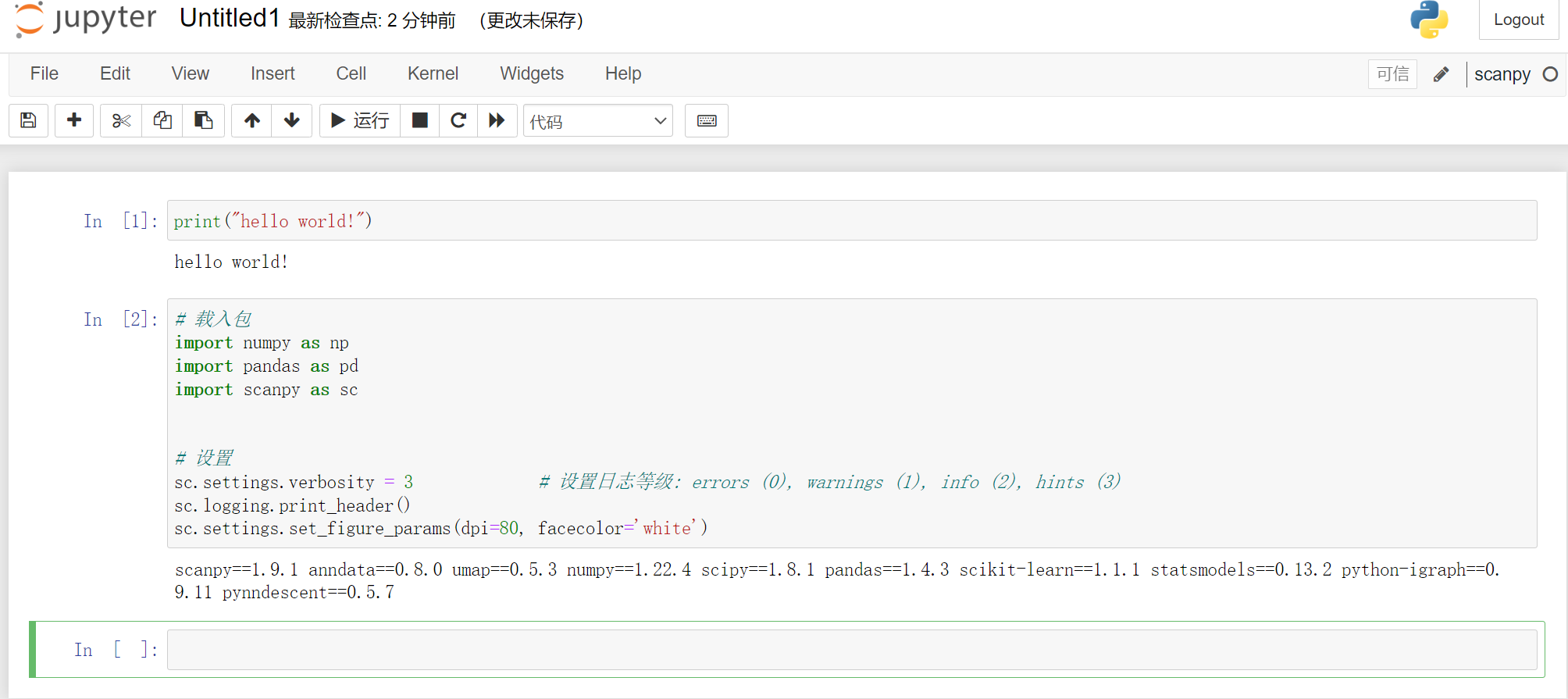
来看看是否能够运行,输入
print("hello world!")
# 载入包
import numpy as np
import pandas as pd
import scanpy as sc
# 设置
sc.settings.verbosity = 3 # 设置日志等级: errors (0), warnings (1), info (2), hints (3)
sc.logging.print_header()
sc.settings.set_figure_params(dpi=80, facecolor='white')# 载入包
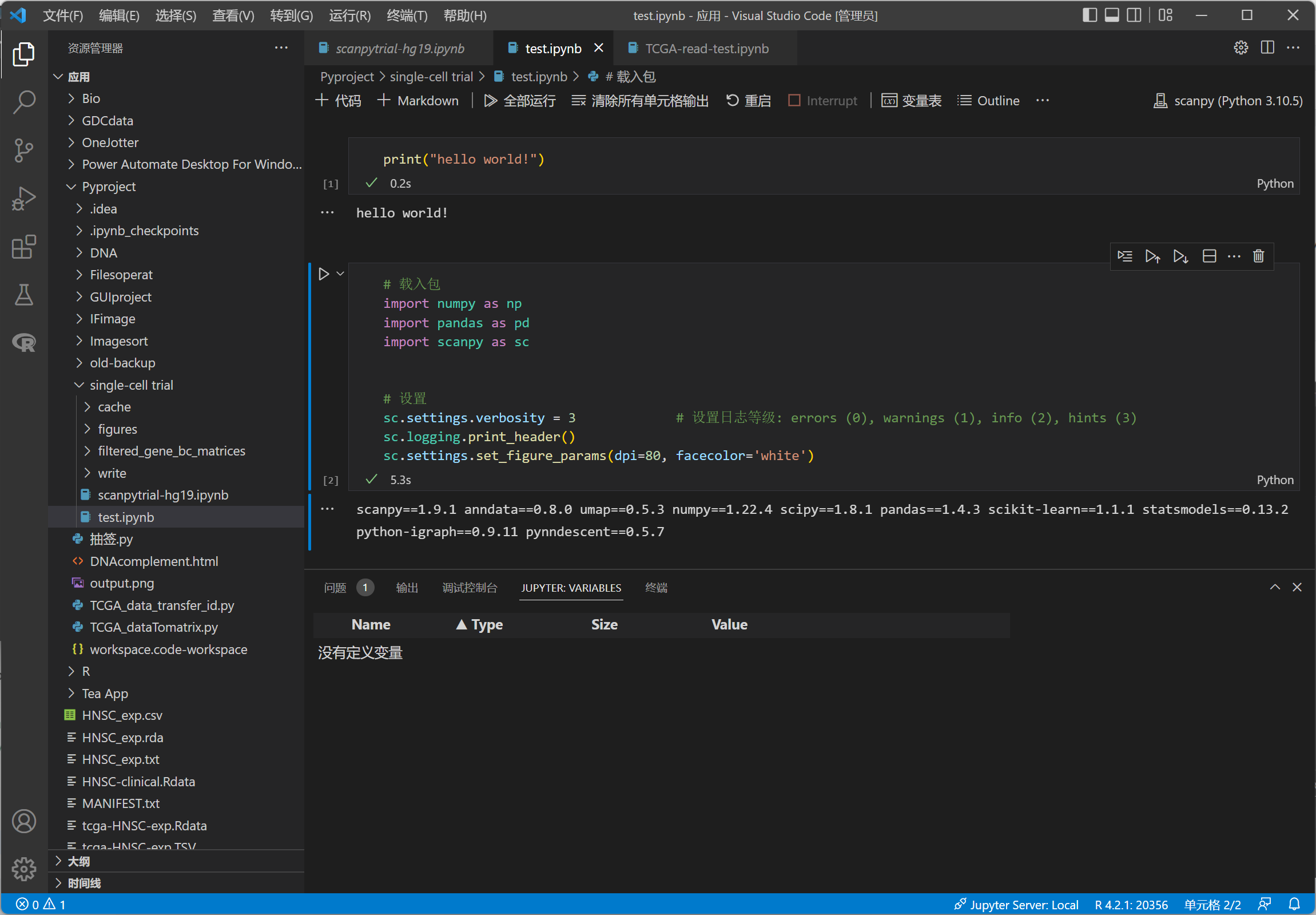
- 再来看看VSCode
最后
到这里就能够使用scanpy进行分析了,具体的分析操作可以查阅 Scanpy Tutorials 进行学习。
下一次,我会分享在单细胞分群中遇到的一些问题以及解决方法。

